
ページ読込中...

ページ読込中...

[English]
| 木下 一彦 [教授] |  |
|
| homepage | http://www.k2.phys.waseda.ac.jp | |
| 専門分野 | 生物物理学 | |
| 研究テーマ・研究活動 | ||
| ○光学顕微鏡下の一分子生理学 ○たんぱく質分子機械の働く仕組み |
||
| ●日本生物物理学会 ●日本物理学会 ●日本細胞生物学会 ●アメリカ生物物理学会 |
||
| 編著: 「蛍光測定-生物科学への応用」(学会出版センター)、 「限界を越える生物顕微鏡-見えないものを見る」(学会出版センター) |
||
肉や魚でおなじみのたんぱく質、私たちの体を作っているたんぱく質は、すべての物質同様、もとをただせば分子です。たんぱく質の分子は、たった1個だけで、あたかも機械のように見事な機能を発揮するので、「分子機械」と呼ばれます。私たちが考えたり、走ったり、見たり、食べたりするのは、みんな分子機械の働きのおかげです。たとえば食べ物からエネルギーを取り出す分子機械は、何とぐるぐる回りながら、ATPと呼ばれるエネルギー源物質を作り出します。脳の中で栄養を運ぶのは、2本足のリニアーモーターです。これら分子機械の大きさは、10ナノメートル程度、原子が数十個並ぶだけです。こんな小さな分子機械、生き物の中にある天然のナノマシンが、いったいどうやって働くのか、その仕掛けを探りたいのです。
生体分子機械は、「機械のように精確に」退屈なリズムを刻んで動くわけではなく、ヒトのように気まぐれや間違いをまじえて働きます。光学顕微鏡で覗いてやると、生きているとしか思えない分子の振るまいが、目の前に見えてきます。分子のような小さなものがどうして顕微鏡で見えるか。そこは工夫をするのです。分子機械は力持ちなので、自分よりはるかに大きな目印をくっつけられても平気で動けます。顕微鏡で見えるような長い棒をつけてやれば、分子機械の微妙な動きを棒が拡大して見せてくれます。あるいは、分子機械の特定の場所にぴかっと光る目印を付けます。大きな目印は光や磁石を使って外から動かせるので、ナノメートルサイズの分子機械を引っ張ったりひねったりもできるのです。1個1個をじっくり観察し、いじってみて、働き方を探る。ヒトや動植物の代わりに分子を相手の生理学、「一分子生理学」です。
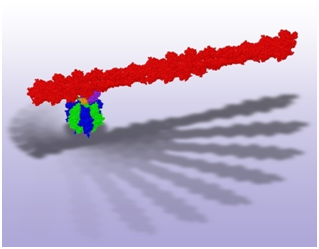 体の中でくるくる回る分子モーターF1(えふわん)。緑3つ、青3つで出来た筒の中でオレンジの棒が回ります。それを光学顕微鏡で見るために、紫の分子を糊にして、赤で示した棒(アクチンというたんぱく質分子がつながったもの)を付けてやりました。クルクルと左回りするのが見えました(研究室ホームページにビデオあり)。粒の一つ一つは原子です。
体の中でくるくる回る分子モーターF1(えふわん)。緑3つ、青3つで出来た筒の中でオレンジの棒が回ります。それを光学顕微鏡で見るために、紫の分子を糊にして、赤で示した棒(アクチンというたんぱく質分子がつながったもの)を付けてやりました。クルクルと左回りするのが見えました(研究室ホームページにビデオあり)。粒の一つ一つは原子です。
| Kazuhiko Jr Kinoshita [Professor] |  |
|
| homepage | http://www.k2.phys.waseda.ac.jp | |
| research field | Single-molecule physiology: How protein machines may work | |
| research keywords | ||
| Seeing-is-understanding type single-molecule physiology Mechanism of rotary molecular motor/generator F1-ATPase Mechanisms of linear molecular motors |
||
| link | ||
A single molecule of a protein (or RNA) enzyme acts as a machine that carries out a unique function in cellular activities. These molecular machines, unlike man-made machines, operate stochastically and thus cannot be synchronized with each other.
To elucidate their mechanisms, therefore, one needs to observe closely the behaviors of individual molecules. Our preference is to attach to the nanometer-sized molecular machines a probe that is huge compared to the machine size, such as a micron-sized plastic bead. Watching the motion of a huge probe is easy, allowing seeing-is-understanding type experiments under an optical microscope. For example, that F1-ATPase is a rotary molecular motor was convincingly demonstrated through the rotary motion of a micron-long actin filament attached to the putative rotor subunit (image below) [Nature 386 (1997) 299-302]. A huge probe also serves as a handle for manipulation, with optical or magnetic tweezers.
We have been able to show that F1-ATPase rotates in steps of 80°and 40°[Nature 410 (2001) 898-904] and how these steps are driven by the chemical reactions (ATP hydrolysis) in three catalytic sites in the stator portion [Cell 130 (2007) 309-321]. F1 is a powerful motor with energy-conversion efficiency possibly approaching 100% [Cell 93 (1998) 1117-1124]. When we forcibly let this motor rotate in reverse with magnetic tweezers, it synthesized ATP, showing that F1 is a completely reversible molecular machine [Nature 427 (2004) 465-468]. An astonishing puzzle, yet unsolved, is that rotation of F1 does not require its axle [Science 319 (2008) 955-958].
RNA polymerase, a machine that reads the genetic codes on DNA, has been shown to spiral around the double helix of DNA, like a nut on a bolt [Nature 409 (2001) 113-115]. In contrast, a linear molecular motor myosin V proceeds in a left-handed spiral around the right-handed helix of actin, indicating that it ‘walks’ with discrete steps, rather than slides, along actin [Nat. Struct. Biol. 9 (2002), 464-467]. It does walk, but, unlike a human, a lifted leg is swirled in all directions by Brownian motion, implying that the leg-leg junction is a completely free joint [Science 316 (2007) 1208-1212]. A fun game has been to tie a knot in DNA with optical tweezers [Nature 399 (1999) 446-448].
Our primary interest has been on chemo-mechanical energy converters that use the free energy of nucleotide hydrolysis to drive mechanical motion. We aim at presenting general principle(s) of operation for these machines. Best understood is the F1-ATPase, which seems to work by a series of induced fit and induced ‘unfit,’ or conformational changes accompanying substrate binding and product release. Conversely, the conformational changes, driven thermally or by an external force, modulate the affinity for substrates and products. The interplay between conformation and affinity changes is the key for chemo-mechanical coupling and, in F1-ATPase, for the reverse process of mechano-chemical coupling.
 F1-ATPase, a part of the enzyme ATP synthase, consists of six stator subunits (green and blue) and a central rotor subunit (orange). Its rotation, powered by ATP hydrolysis, has been videotaped under an optical microscope by attaching a long actin filament (red; actual length far exceeds the image size) to the rotor through streptavidin (purple).
F1-ATPase, a part of the enzyme ATP synthase, consists of six stator subunits (green and blue) and a central rotor subunit (orange). Its rotation, powered by ATP hydrolysis, has been videotaped under an optical microscope by attaching a long actin filament (red; actual length far exceeds the image size) to the rotor through streptavidin (purple).